Variabilidad de la secuencia Pr77 del retrotransposón L1Tc.
Inmaculada Gómez, Manuel Carlos López, Alberto Rastrojo, Fabian Lorenzo-Díaz, Jose María Requena, Begoña Aguado, Basilio Valladares, M. Carmen Thomas
Acta Tropica, Volumen 222, Octubre 2021.
RESUMEN
Todos los genomas de tripanosomátidos están colonizados por retrotransposones sin LTR que portan en sus extremos 5 ' una secuencia de 77 nucleótidos altamente conservada denominada "huella Pr77" o "sello Pr77". La huella Pr77 tiene doble función, a nivel de ADN posee actividad promotora y, a nivel de ARN, tiene función ribozima de tipo HDV-like (Hepatitis Delta Virus) por lo que es esperable que, su extensa distribución, esté relacionada con los procesos de regulación génica en estos organismos.
En este trabajo se reporta la identificación de retrotransposones portadores del sello Pr77 (L1Tc, NARTc, DIRE) y el análisis de las asociaciones de elementos móviles con genes relevantes en los genomas de seis cepas de Trypanosoma cruzi, aisladas de diferentes hospedadores y vectores, las cuales pertenecen a diferentes unidades de tipificación discreta (DTU o Discrete Type Unit). Para ello, se han secuenciado, ensamblado y anotado los genomas de seis cepas [B. M. Lopez (TcIa), Dm28 (TcId), Y (TcII), Ikiakarora (TcIII), SOL (TcV) y CL Brener (TcVI)] de este parásito humano. Los análisis BUSCO realizados indicaron una buena calidad de los ensamblajes realizaros los cuales se utilizaron en los análisis comparativos. Los resultados obtenidos muestran diferencias entre los seis genomas en cuanto al número de copias de genes relacionados con los procesos de virulencia, la abundancia de retrotransposones que llevan la huella Pr77 así como la presencia de la huella Pr77 no asociada a retroelementos. Los análisis muestran también asociaciones frecuentes de retrotransposones portadores de Pr77 así como de huellas Pr77 no asociadas a retroelementos con genes que codifican trans-sialidasas, RHS, MASP o proteínas de función no conocida, mostrando una proporción variable según el tipo de retroelemento, la clase de genes y la cepa del parásito. Estas diferencias en la distribución genómica de retroelementos activos y otros elementos que contienen Pr77 han dado forma a la arquitectura del genoma de estas seis cepas y podrían estar contribuyendo a la variabilidad fenotípica existente entre ellas. Dado que la secuenciación de estos genomas contribuye al actual repositorio de T. cruzi, el hallazgo aquí reportado destaca la variabilidad entre las diferentes cepas de este complejo parásito en el que la huella Pr77 parece tener una funcional relevante.
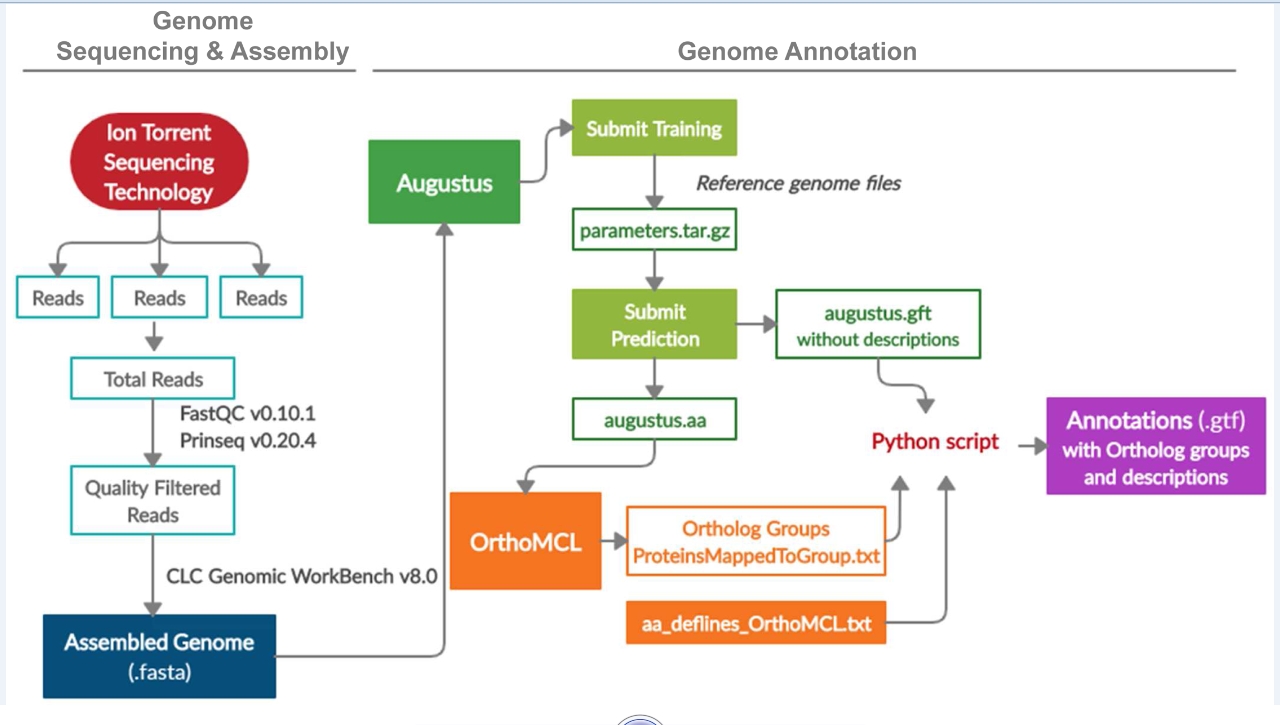
Fig: Diagrama esquemático que describe los procesos de secuenciación, ensamblaje y anotación llevados a cabo para generar los ensamblajes genómicos y anotaciones de los genomas de las diferentes cepas de Trypanosoma cruzi descritas en este estudio.
https://doi.org/10.1016/j.actatropica.2021.106053
